OpenCVの使い方28 ~ 輪郭抽出応用
今回は前回までのOpenCVを用いた輪郭抽出の応用例として赤血球のカウント、サイズ分布について紹介したい。
1. 画像読み込みと2値化処理
今回は適当な赤血球の顕微鏡画像を用意してGoogle Corabで読み込んだものを使用する。
Google Colabでのサンプル画像読み込み
import cv2 import numpy as np from matplotlib import pyplot as plt from google.colab import files uploaded_file = files.upload() uploaded_file_name = next(iter(uploaded_file)) orig = cv2.imread(uploaded_file_name)
もとのカラー画像をsrc, 2値化処理のためのグレー画像をgrayとして格納する。
src = cv2.cvtColor(orig, cv2.COLOR_BGR2RGB)
gray = cv2.cvtColor(orig, cv2.COLOR_BGR2GRAY)
plt.imshow(src)
plt.title('Original')
plt.imshow(gray,cmap='gray') plt.title('Gray')
次に2値化処理をするが、暗い部分が赤血球になり、今回は暗い部分を白にするためcv2.thresholdのオプションをcv2.THRESH_BINARY_INVと指定する。
ret,thresh = cv2.threshold(gray,110,255,cv2.THRESH_BINARY_INV) plt.imshow(thresh,cmap='gray',vmin=0,vmax=255) plt.title('Binary')

閾値を110としたが、数値を変えると輪郭抽出の結果が異なってくる。
2. 赤血球輪郭の抽出
2値化したバイナリ画像からcv2.findContours()を用いて輪郭を抽出する際、赤血球内部の輪郭は不要のため、外部のみの輪郭を抽出するcv2.RETR_EXTERNALを指定する。
contours, hierarchy = cv2.findContours(thresh,cv2.RETR_EXTERNAL,cv2.CHAIN_APPROX_SIMPLE) cnt_img = np.zeros_like(gray, dtype=np.uint8) cnt_img_tree = cv2.drawContours(cnt_img, contours, -1, 255, 2) plt.imshow(cnt_img_tree,cmap='gray',vmin=0,vmax=100)

抽出した輪郭がバイナリ画像とあっているか確認するため、バイナリ画像の強度を下げて輪郭画像と重ね合わせる。
th2=(thresh/10) plt.imshow(cnt_img_tree+th2,cmap='gray',vmin=0,vmax=100)
おおざっぱに輪郭抽出が正しいことが確認できる。

3. サイズ分布
抽出した輪郭の数から赤血球をカウントするが、画像と比較してかなり多いことがわかる。
n=len(contours) n >624
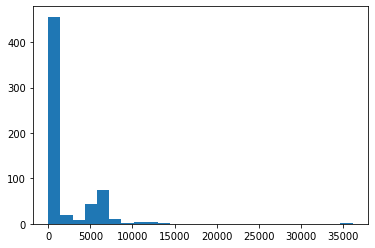
そこで輪郭の面積をcv2.contourArea()で取得し、ヒストグラムをプロットしてサイズ分布を確認する。
ind=[] areas=[] for i in np.arange(n): cnt=contours[i] area = cv2.contourArea(cnt) ind.append(i) areas.append(area) plt.hist(areas,bins=25)
明らかに面積が小さいものが偏って含まれており、画像のノイズと考えられる。また大きいサイズのものは赤血球が2つ以上重なっているものと考えられる。
4. イレギュラーな輪郭の除外
イレギュラーなものを除外した赤血球のサイズ分布を調べる。ノイズと赤血球が重なったものを除外するためサイズを3500以上8500以下のものに限定する。
ind=[] areas=[] for i in np.arange(n): cnt=contours[i] area = cv2.contourArea(cnt) if area > 3500 and area<8500: ind.append(i) areas.append(area) n2=np.size(ind) n2 >130
サイズを限定することで抽出した輪郭数が130まで減少した。
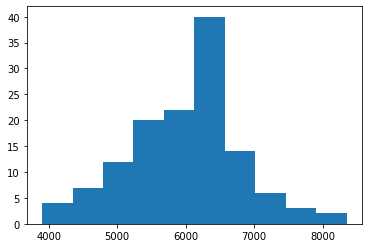
plt.hist(areas)
サイズの分布は6000強をピークとする分布になっていることがわかる。
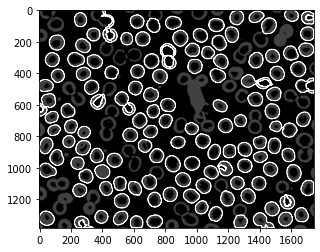
サイズ制限した場合の輪郭とバイナリ画像を重ね合わせると、概ね2つ以上重なっているものは除外できていることが確認できる。また画像端にある赤血球の輪郭も除外されていることがわかる。またカウントとしては重なっているものを除外したため精度はよいとは言えないが、例えばサイズ制限で通常の2倍程度のものを抽出するなどの工夫の余地はある。
img2 = np.zeros_like(thresh, dtype=np.uint8) for i in np.arange(n2): cnt_img2 = np.zeros_like(thresh, dtype=np.uint8) cnt_img2 = cv2.drawContours(cnt_img2, contours,ind[i], 255, 2) img2=img2+cnt_img2 plt.imshow(img2+th2,cmap='gray',vmin=0,vmax=100)
5. まとめ
今回は輪郭抽出の応用として実際の赤血球の画像からカウントとサイズ分布を調べる方法について紹介した。